

--> Download : Cliquez sur ce Lien (FIJI : Fiji Is Just ImageJ !!) pour récupérer l'Appli ImageJ correspondant à votre machine.
Table des matières (à cliquer):
I. Etude d’un gel d’électrophorèse :
1. Obtention du densitogramme du gel :
2. Calibration des distances de migration :
3. Mesure des distances de migration des marqueurs de paires de bases :
4. Réalisation de la courbe étalon et détermination des tailles des fragments inconnus :
II. Dénombrement et analyse d’un ensemble de particules : (Ex : Bactéries)
1. Dénombrement de particules de même taille :
2. Dénombrement de particules de forme différente et de couleur différente :
3. Dénombrement de particules de même taille mais de couleur différente :
4. Sélection et Dénombrement de particules selon la taille :
III. Dosage colorimétrique sans spectrophotomètre : Macros & Shorcuts.
1. Tests immunologique (type ELISA) sur microplaque.
2. Dosage colorimétrique sur cuves spectrophotométriques : gamme d’étalonnage et essai :
I. Etude d’un gel d’électrophorèse :
1. Obtention du densitogramme du gel :
2. Calibration des distances de migration :
3. Mesure des distances de migration des marqueurs de paires de bases :
4. Réalisation de la courbe étalon et détermination des tailles des fragments inconnus :
II. Dénombrement et analyse d’un ensemble de particules : (Ex : Bactéries)
1. Dénombrement de particules de même taille :
2. Dénombrement de particules de forme différente et de couleur différente :
3. Dénombrement de particules de même taille mais de couleur différente :
4. Sélection et Dénombrement de particules selon la taille :
III. Dosage colorimétrique sans spectrophotomètre : Macros & Shorcuts.
1. Tests immunologique (type ELISA) sur microplaque.
2. Dosage colorimétrique sur cuves spectrophotométriques : gamme d’étalonnage et essai :
1) Obtention du densitogramme du gel :
· Ouvrir ImageJ : Le logiciel s’ouvre sous la forme d’une barre d’outils :
· Ouvrir ImageJ : Le logiciel s’ouvre sous la forme d’une barre d’outils :
· Cliquer gauche sans relacher sur le nom de fichier correspondant à la photographie du gel à étudier « Gel ADN », et le faire glisser jusqu'à la barre grise sous les pictogrammes de la barre d'outils ;
o Recommandations : cette photographie devra présenter des pistes d’électrophorèse parfaitement verticale et contenir un repère étalon de distance de migration.
· A l’aide de l’outil « Rectangular » , dessiner un rectangle encadrant la piste d’électrophorèse contenant les marqueurs, en démarrant exactement au niveau de la base du puits d’électrophorèse :
o Recommandations : cette photographie devra présenter des pistes d’électrophorèse parfaitement verticale et contenir un repère étalon de distance de migration.
· A l’aide de l’outil « Rectangular » , dessiner un rectangle encadrant la piste d’électrophorèse contenant les marqueurs, en démarrant exactement au niveau de la base du puits d’électrophorèse :
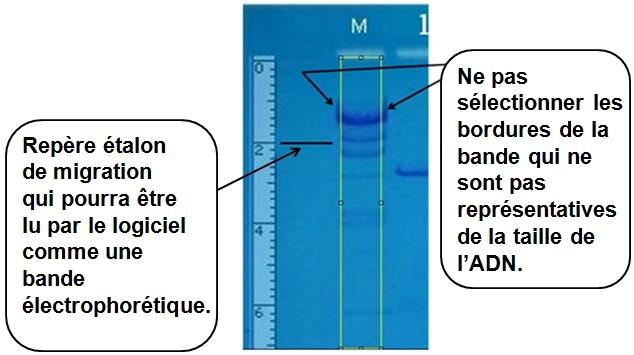
Ce qu’il faut comprendre c’est que ce rectangle servira de sonde de lecture pour toutes les pistes ; En aucun cas, ImageJ acceptera de faire des analyses sur plusieurs pistes avec des rectangles différents !
· Cliquer ensuite dans la barre d’outils, sur « Analyse »/« Gels »/«Select First Lane ».
Il apparait alors un petit chiffre « 1 » en blanc au centre de notre piste d’électrophorèse encadrée.
· Copier (Ctrl C) ce rectangle.
· Coller (Ctrl V) ce rectangle sur cette photographie : Attention l’objet collé contient exactement l’image de la piste copiée. Il nous faut donc éliminer l’image contenue dans ce cadre tout en conservant le cadre.
· Faire Ctrl Z pour éliminer l’image contenue dans ce cadre collé.
· Ensuite, déplacer ce cadre sous la piste d’électrophorèse «1 » (Faire en sorte que les rectangles soient parfaitement alignés horizontalement).
· Enfin, cliquer sur « Analyse »/« Gels »/«Select Next Lane ».
Il apparait alors un petit chiffre « 2 » en blanc au centre de notre piste d’électrophorèse encadrée.
· Répéter les étapes précédentes jusqu’à ce que toutes les pistes aient été encadrées et identifiées par un chiffre.
· Ensuite cliquer sur « Analyse »/« Gels »/« Plot Lanes » :
· Cliquer ensuite dans la barre d’outils, sur « Analyse »/« Gels »/«Select First Lane ».
Il apparait alors un petit chiffre « 1 » en blanc au centre de notre piste d’électrophorèse encadrée.
· Copier (Ctrl C) ce rectangle.
· Coller (Ctrl V) ce rectangle sur cette photographie : Attention l’objet collé contient exactement l’image de la piste copiée. Il nous faut donc éliminer l’image contenue dans ce cadre tout en conservant le cadre.
· Faire Ctrl Z pour éliminer l’image contenue dans ce cadre collé.
· Ensuite, déplacer ce cadre sous la piste d’électrophorèse «1 » (Faire en sorte que les rectangles soient parfaitement alignés horizontalement).
· Enfin, cliquer sur « Analyse »/« Gels »/«Select Next Lane ».
Il apparait alors un petit chiffre « 2 » en blanc au centre de notre piste d’électrophorèse encadrée.
· Répéter les étapes précédentes jusqu’à ce que toutes les pistes aient été encadrées et identifiées par un chiffre.
· Ensuite cliquer sur « Analyse »/« Gels »/« Plot Lanes » :

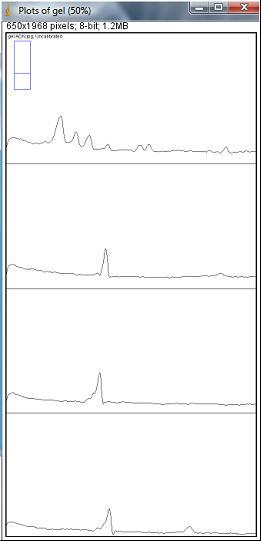
un nouveau cadre d’image apparait : il contient, empilés les uns sur les autres, l’ensemble des densitogrammes souhaités.
Pour voir tous les densitogrammes, il faut se placer sur chacun d’entre eux et dézoomer (taper sur la touche -) ou zoomer (taper sur la touche +).
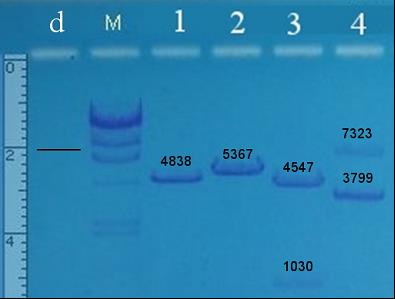
De cette électrophorèse, on ne connait que la taille des ADN marqueurs (23129, 9416, 6557, 4361, 2322, 2027, et 564 paires de bases). On ne connait pas les tailles des ADN migrant sur les pistes 1 à 5, et c’est ce que l’on veut découvrir.
Pour voir tous les densitogrammes, il faut se placer sur chacun d’entre eux et dézoomer (taper sur la touche -) ou zoomer (taper sur la touche +).
De cette électrophorèse, on ne connait que la taille des ADN marqueurs (23129, 9416, 6557, 4361, 2322, 2027, et 564 paires de bases). On ne connait pas les tailles des ADN migrant sur les pistes 1 à 5, et c’est ce que l’on veut découvrir.
2) Calibration des distances de migration :
· Enregistrer le densitogramme.
Puis le fermer et l’ouvrir pour l’analyser avec ImageJ.
· Zoomer sur le dernier densitogramme (piste d).
· Sélectionner l’icône « Straight » : ;
· Placez alors le curseur de la souris quasiment au sommet du pic de ce densitogramme « d », Tout en maintenant enfoncé la touche Maj (permet de tracer une ligne horizontale),
cliquer gauche sans relâcher, puis dirigez la souris vers la marge de gauche : un trait horizontal avec 3 petits carrés est dessiné :
· Enregistrer le densitogramme.
Puis le fermer et l’ouvrir pour l’analyser avec ImageJ.
· Zoomer sur le dernier densitogramme (piste d).
· Sélectionner l’icône « Straight » : ;
· Placez alors le curseur de la souris quasiment au sommet du pic de ce densitogramme « d », Tout en maintenant enfoncé la touche Maj (permet de tracer une ligne horizontale),
cliquer gauche sans relâcher, puis dirigez la souris vers la marge de gauche : un trait horizontal avec 3 petits carrés est dessiné :
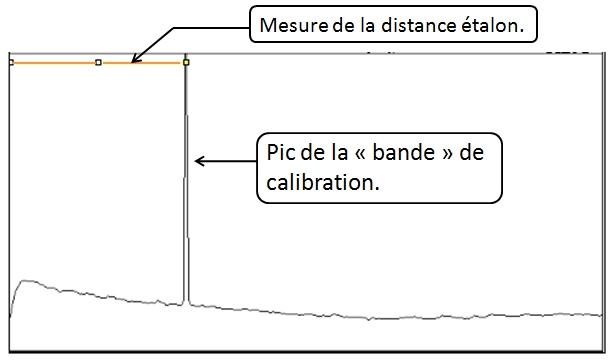
· Faîtes : « Analyse »/ « Set Scale » ;
Dans « Known distance », écrire 200 ;
Dans « Unit » : écrire « mm », puis faire OK.
· Cliquer sur « Analyse »/ « Set measurements », une fenêtre s’ouvre : tout désélectionner et mettre un 0 dans le « Decimal Places ». Puis faire Ok.
3) Mesure des distances de migration des bandes électrophorétiques :
· Dézoomer jusqu’à apercevoir le 1er densitogramme (piste marqueurs) et zoomer dessus.
· Placer le curseur de la souris au sommet du 1er pic, maintenir la touche Maj enfoncée et maintenir le clic gauche jusqu’à atteindre la marge de gauche de l’image, puis relâcher.
· Faire Ctrl M : un tableau s’ouvre mentionnant le N° du pic étudié et la migration en mm (Length= distance) :
Dans « Known distance », écrire 200 ;
Dans « Unit » : écrire « mm », puis faire OK.
· Cliquer sur « Analyse »/ « Set measurements », une fenêtre s’ouvre : tout désélectionner et mettre un 0 dans le « Decimal Places ». Puis faire Ok.
3) Mesure des distances de migration des bandes électrophorétiques :
· Dézoomer jusqu’à apercevoir le 1er densitogramme (piste marqueurs) et zoomer dessus.
· Placer le curseur de la souris au sommet du 1er pic, maintenir la touche Maj enfoncée et maintenir le clic gauche jusqu’à atteindre la marge de gauche de l’image, puis relâcher.
· Faire Ctrl M : un tableau s’ouvre mentionnant le N° du pic étudié et la migration en mm (Length= distance) :

· Continuer ainsi sur tous les pics de la piste étalon, puis sur les pics des pistes 1 à 4.
· Sauvegarder le tableau en cliquant sur « File »/ « Save As ».
4) Réalisation de la courbe étalon et détermination des tailles des fragments inconnus :
· Fermer, puis ouvrir le tableau : par défaut, Excel récupère toutes les données d’ImageJ.
· Ajouter une colonne « Nombre de paire de base » et une autre colonne « Log du nombre de paire de base ».
· Remplir correctement ces deux colonnes.
· Réaliser le graphique Log (pb) = f (migration) en demandant une courbe de tendance linéaire :
· Sauvegarder le tableau en cliquant sur « File »/ « Save As ».
4) Réalisation de la courbe étalon et détermination des tailles des fragments inconnus :
· Fermer, puis ouvrir le tableau : par défaut, Excel récupère toutes les données d’ImageJ.
· Ajouter une colonne « Nombre de paire de base » et une autre colonne « Log du nombre de paire de base ».
· Remplir correctement ces deux colonnes.
· Réaliser le graphique Log (pb) = f (migration) en demandant une courbe de tendance linéaire :
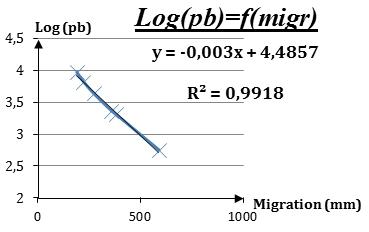
· Utiliser la formule de tendance linéaire pour obtenir le Log(pb) des fragments de taille inconnue en fonction de leur distance de migration.
· transformer ces Log(pb) des fragments inconnus en nombre de paire de bases :
· transformer ces Log(pb) des fragments inconnus en nombre de paire de bases :
II Dénombrement et analyse d’un ensemble de colonies bactériennes :
Problématique :
Dénombrer un ensemble de colonies bactériennes en fonction :
Dénombrer un ensemble de colonies bactériennes en fonction :
- De la forme,
- Des couleurs,
- De la taille.
1) Dénombrement de colonies bactériennes de même taille :
· Cliquer gauche sans relacher sur le nom de fichier correspondant à la photographie du gel à étudier « Dénombrement 1 », et le faire glisser jusqu'à la barre grise sous les pictogrammes de la barre d'outils :
· Cliquer gauche sans relacher sur le nom de fichier correspondant à la photographie du gel à étudier « Dénombrement 1 », et le faire glisser jusqu'à la barre grise sous les pictogrammes de la barre d'outils :

Nous allons tout d’abord donner à ImageJ l’échelle de cette image ;
· Cliquer sur l’ icône : « Straight » : , pour dessiner une ligne en bas à droite de l’image, d’une longueur d’environ 3 petites colonies bactériennes.
une ligne avec 3 petits carrés s’affiche :
· cliquer ensuite sur « Analyse »/ « Set scale » : une fenêtre s’ouvre ; renseigner comme suit : « Known distance » : 1.00 ; « Unit of length » : cm. Puis OK.
· Puis faire « Edit »/ « Selection »/ « Select none » : pour retirer ce tracé.
Ensuite, il nous faut séparer les éléments visuels présents sur la photo :
· Cliquer sur « Image »/ « Adjust »/ « Color Treshold » : une fenêtre nommée « Treshold color » apparait, tandis que notre photo a été colorée.
· Cliquer sur l’ icône : « Straight » : , pour dessiner une ligne en bas à droite de l’image, d’une longueur d’environ 3 petites colonies bactériennes.
une ligne avec 3 petits carrés s’affiche :
· cliquer ensuite sur « Analyse »/ « Set scale » : une fenêtre s’ouvre ; renseigner comme suit : « Known distance » : 1.00 ; « Unit of length » : cm. Puis OK.
· Puis faire « Edit »/ « Selection »/ « Select none » : pour retirer ce tracé.
Ensuite, il nous faut séparer les éléments visuels présents sur la photo :
· Cliquer sur « Image »/ « Adjust »/ « Color Treshold » : une fenêtre nommée « Treshold color » apparait, tandis que notre photo a été colorée.
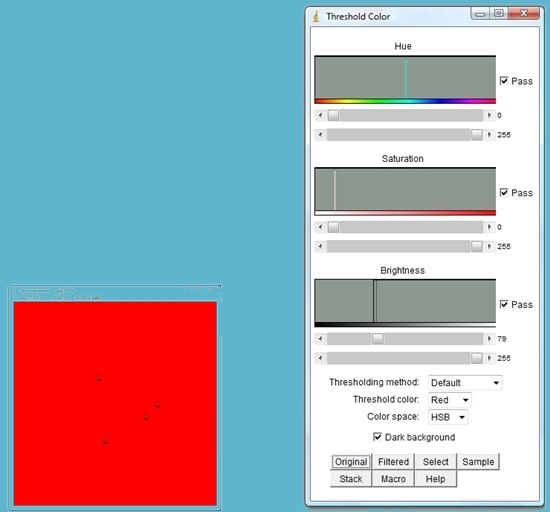

· Dans la fenêtre Treshold color, cliquer, tout en bas sur « Original » : la photo, dans sa configuration initiale réapparait ;
· Dans le menu d’icônes d’ImageJ, sélectionner l’icône « Oval » ; après avoir zoomé sur une colonie, dessiner une sphère à l’intérieur.
· Puis cliquer, dans la fenêtre Treshold color, sur « Sample » : dès lors, les 4 colonies bactériennes se colorent en rouge !
· Dans le menu d’icônes d’ImageJ, sélectionner l’icône « Oval » ; après avoir zoomé sur une colonie, dessiner une sphère à l’intérieur.
· Puis cliquer, dans la fenêtre Treshold color, sur « Sample » : dès lors, les 4 colonies bactériennes se colorent en rouge !
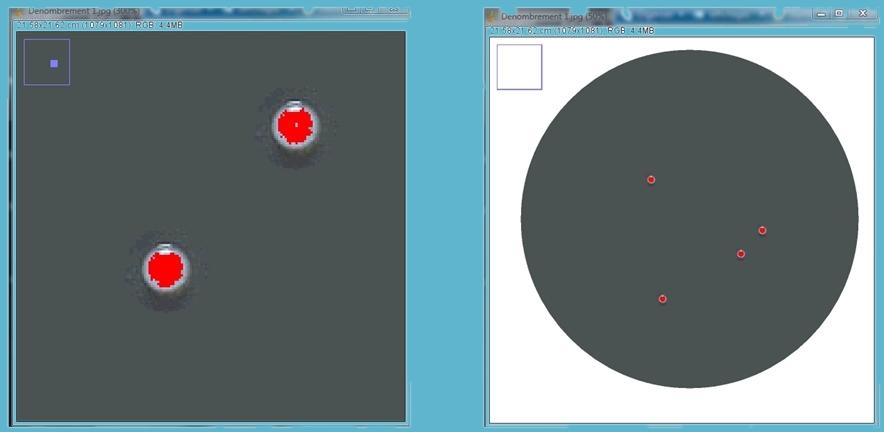
Il nous reste plus qu’à demander au logiciel de compter ce qui a été coloré.
· Cliquer dans le menu sur « Analyse »/ « Analyse particles » ;
Une fenêtre « Analyse particles » s’ouvre,
· Cliquer dans le menu sur « Analyse »/ « Analyse particles » ;
Une fenêtre « Analyse particles » s’ouvre,
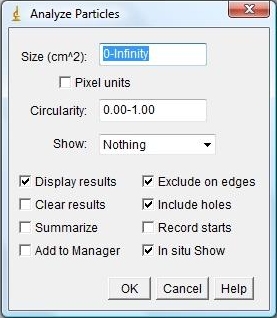
Elle contient :
o Size : Remarquez les unités : cm² : 0 – Infinity : dans cette configuration, le logiciel va donc chercher des particules qui auront une aire supérieure à 0 cm² sans limite supérieure :
Pour cette photographie, on ne modifie pas ce paramètre, mais c’est ici que l’on pourra agir si l’on souhaite compter des particules de tailles différentes.
o Circularity : 0.00-1.00 : c’est une gamme large de circularité : ainsi le logiciel cherchera des particules non parfaitement circulaires.
o Show : choisir Outlines pour dessiner des pointillés autour des particules comptées.
o Puis cocher Displays results, in situ show ; et faire Ok.
§ La photo se modifie : elle se transforme en dessin des particules dénombrées avec le décompte à l’intérieur (on peut zoomer dessus : décompte de haut en bas !!) ;
§ Un tableau de résultats s’affiche (données enregistrables vers un tableur !) avec pour chaque colonies comptées son aire en cm².
o Size : Remarquez les unités : cm² : 0 – Infinity : dans cette configuration, le logiciel va donc chercher des particules qui auront une aire supérieure à 0 cm² sans limite supérieure :
Pour cette photographie, on ne modifie pas ce paramètre, mais c’est ici que l’on pourra agir si l’on souhaite compter des particules de tailles différentes.
o Circularity : 0.00-1.00 : c’est une gamme large de circularité : ainsi le logiciel cherchera des particules non parfaitement circulaires.
o Show : choisir Outlines pour dessiner des pointillés autour des particules comptées.
o Puis cocher Displays results, in situ show ; et faire Ok.
§ La photo se modifie : elle se transforme en dessin des particules dénombrées avec le décompte à l’intérieur (on peut zoomer dessus : décompte de haut en bas !!) ;
§ Un tableau de résultats s’affiche (données enregistrables vers un tableur !) avec pour chaque colonies comptées son aire en cm².
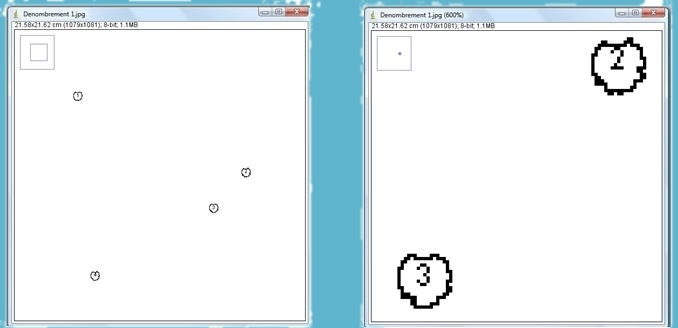
2) Dénombrement de colonies bactériennes de forme différente et de couleur différente :
Le même type de travail que précédemment peut être réalisé sur la photographie « Dénombrement 2 ».
Par contre, pour le dénombrement des colonies de forme lenticulaire, il faudra utiliser l’icône « Freehand Selection » : de manière à bien dessiner la forme.
Le même type de travail que précédemment peut être réalisé sur la photographie « Dénombrement 2 ».
Par contre, pour le dénombrement des colonies de forme lenticulaire, il faudra utiliser l’icône « Freehand Selection » : de manière à bien dessiner la forme.
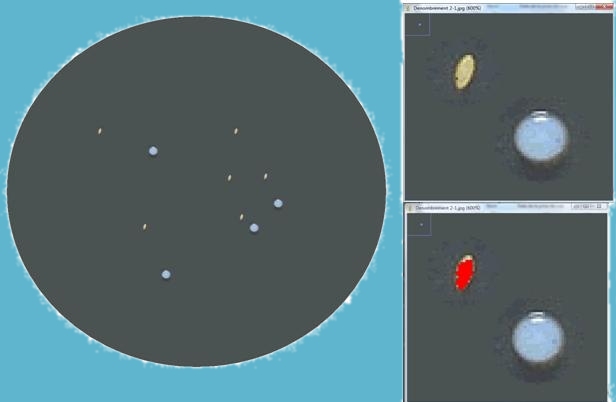
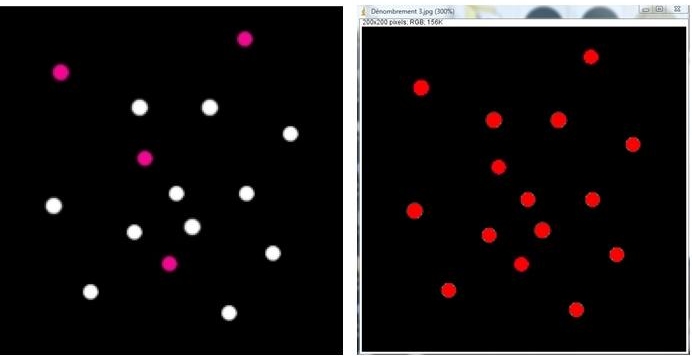
3) Dénombrement de colonies bactériennes de même taille mais de couleur différente :
À présent, nous allons dénombrer en même temps et séparément deux types de particules :
· Cliquer gauche sans relacher sur le nom de fichier correspondant à la photographie du gel à étudier « Dénombrement 3 », et le faire glisser jusqu'à la barre grise sous les pictogrammes de la barre d'outils :
· Passez en Trehold color, et en mode Original ;
· Sélectionner avec le cercle une colonie violette ;
· Puis, maintenez enfoncée la touche Majuscule (Shift – flèche vers le haut) et recommencez une sélection cette fois ci sur une colonie blanche : A présent les deux types de colonies sont sélectionnée.
· Cliquez sur « Sample », et normalement toutes les colonies sont sélectionnées.
· Faire ensuite une analyse de particule ;
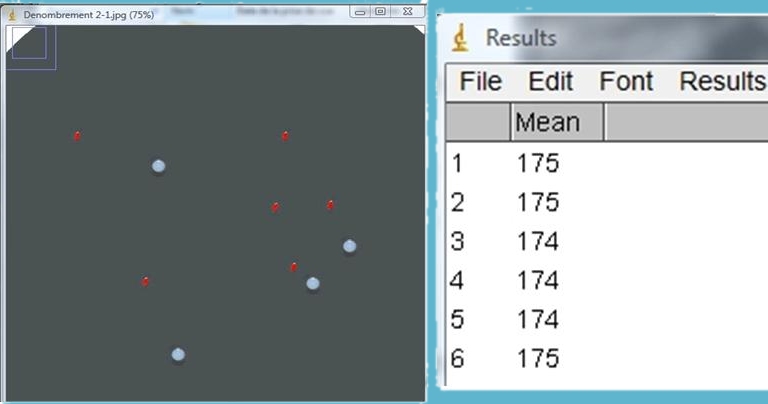
· Le tableau de résultats vous dénombre 15 particules :
À présent, nous allons dénombrer en même temps et séparément deux types de particules :
· Cliquer gauche sans relacher sur le nom de fichier correspondant à la photographie du gel à étudier « Dénombrement 3 », et le faire glisser jusqu'à la barre grise sous les pictogrammes de la barre d'outils :
· Passez en Trehold color, et en mode Original ;
· Sélectionner avec le cercle une colonie violette ;
· Puis, maintenez enfoncée la touche Majuscule (Shift – flèche vers le haut) et recommencez une sélection cette fois ci sur une colonie blanche : A présent les deux types de colonies sont sélectionnée.
· Cliquez sur « Sample », et normalement toutes les colonies sont sélectionnées.
· Faire ensuite une analyse de particule ;
· Le tableau de résultats vous dénombre 15 particules :
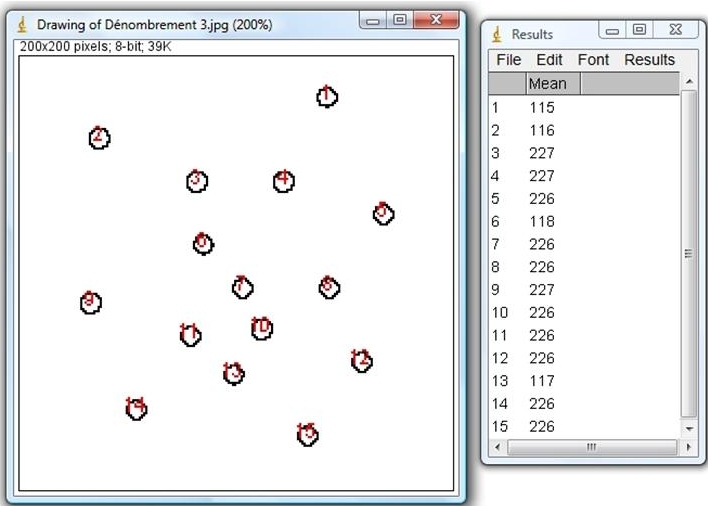
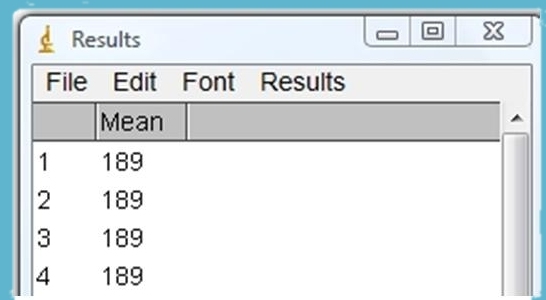
On peut tout remarquer que dans la colonne « Mean » (Nuance de gris moyenne) on observe deux types d’informations : certaines aux alentours de 116, d’autres aux alentours de 227.
On peut donc construire un filtre d’analyse de ce tableau sur Excel.
Pour la colonne « Type de colonie », on pourra utiliser cette formule : =SI(Mean<118;1;2)
Puis, pour le résultat « Nb. de colonies violettes », on utilisera : =NB.SI(Type;1)
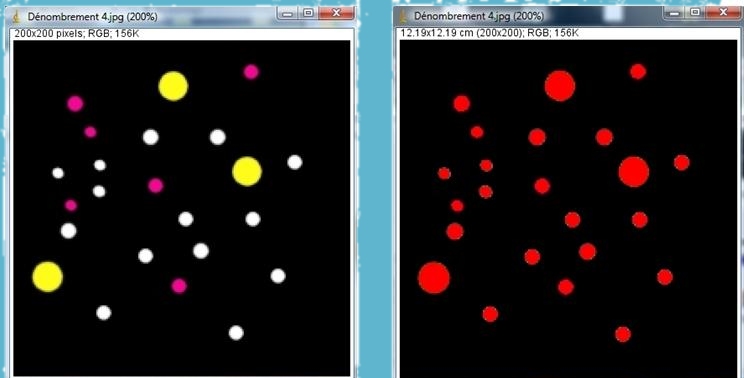
On peut refaire cette analyse avec la photographie « Denombrement 4 » :
On peut donc construire un filtre d’analyse de ce tableau sur Excel.
Pour la colonne « Type de colonie », on pourra utiliser cette formule : =SI(Mean<118;1;2)
Puis, pour le résultat « Nb. de colonies violettes », on utilisera : =NB.SI(Type;1)
On peut refaire cette analyse avec la photographie « Denombrement 4 » :
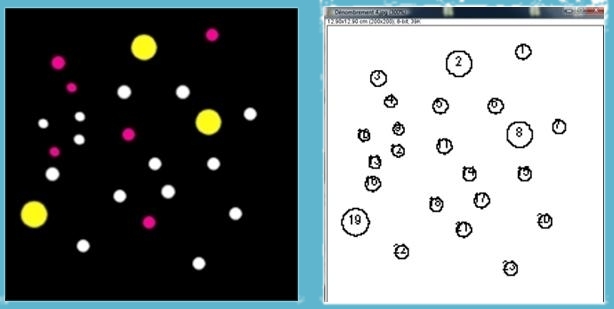

· Refaire cette analyse avec la vraie photographie d’une boîte de Pétri : « Denombrement 5 » :
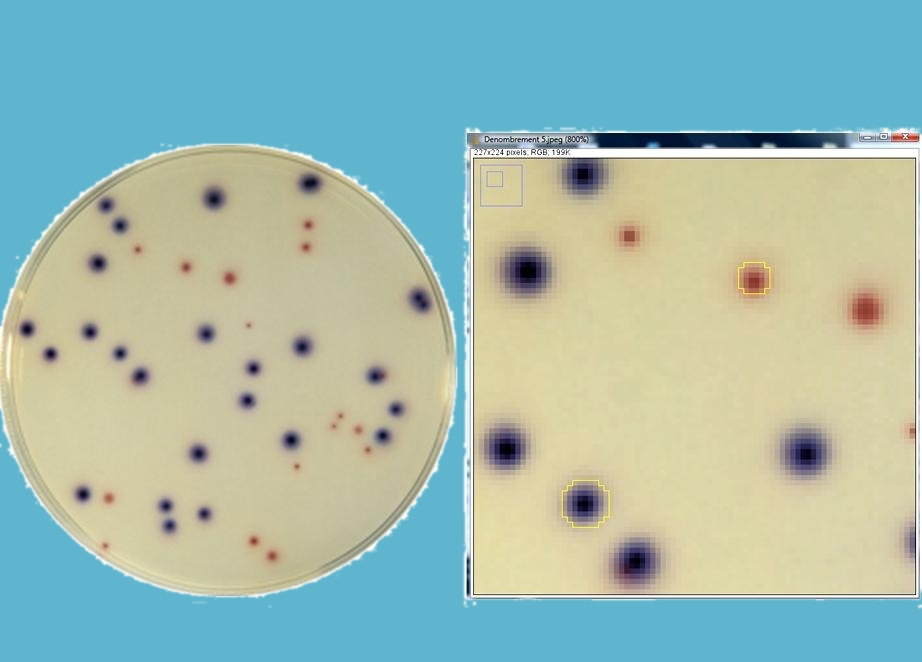
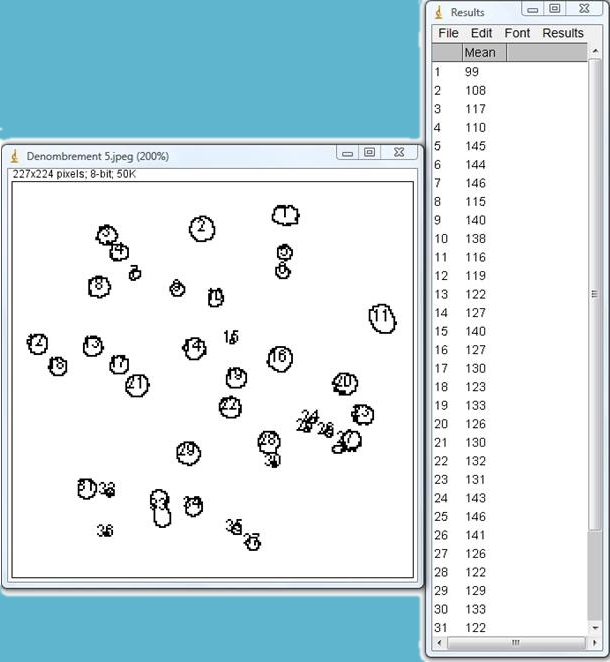
4) Sélection et Dénombrement de colonies bactériennes selon la taille
· Cliquer gauche sans relacher sur le nom de fichier correspondant à la photographie du gel à étudier « Dénombrement 4 », et le faire glisser jusqu'à la barre grise sous les pictogrammes de la barre d'outils :
· Réalisez un étalonnage (Analyse/Set scale) des tailles sur une grosse colonie jaune en indiquant au logiciel qu’elle fait 1 cm ;
· Faire Image/Adjust/Color treshold : le fond de la photo est alors en noir et toutes les colonies sont en rouge ;
· Faire Analyse/Analyse particles :
· Dans size, écrire : 0.70-infinity ; choisir « Show » : Outlines ; cocher Display results et Exclude on edges ; faire OK :
· Cliquer gauche sans relacher sur le nom de fichier correspondant à la photographie du gel à étudier « Dénombrement 4 », et le faire glisser jusqu'à la barre grise sous les pictogrammes de la barre d'outils :
· Réalisez un étalonnage (Analyse/Set scale) des tailles sur une grosse colonie jaune en indiquant au logiciel qu’elle fait 1 cm ;
· Faire Image/Adjust/Color treshold : le fond de la photo est alors en noir et toutes les colonies sont en rouge ;
· Faire Analyse/Analyse particles :
· Dans size, écrire : 0.70-infinity ; choisir « Show » : Outlines ; cocher Display results et Exclude on edges ; faire OK :
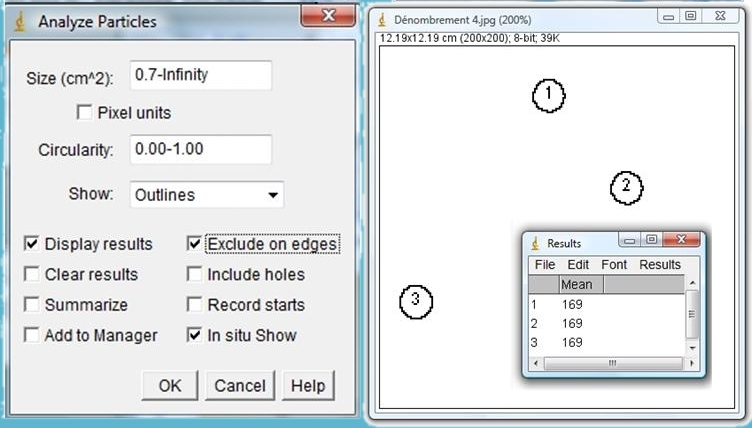
o les trois grosses colonies sont alors comptées
o En effet, on a demandé au logiciel de ne compter que les particules ayant des surfaces comprises entre 0,7 cm² et l’infini.
II Dosage colorimétrique sans spectrophotomètre : Macros & Shorcuts
:
:
1) Tests immunologique (type ELISA) sur microplaque.
Problématique
Réaliser une mesure d’absorbance sans spectrophotomètre : - sur des plaques de 96 puits pour des tests immunologique (type ELISA).
La procédure pour réaliser une lecture optique à l’aide d’ImageJ sans spectrophotomètre n’est pas compliquée, mais lorsqu’elle doit être réalisée sur une plaque de 96 puits, cela devient nettement plus contraignant surtout si cette analyse est à réaliser plusieurs fois par jour en laboratoire.
Ainsi, ImageJ propose à l’utilisateur de créer des Macros : Au moment où on le décide, ImageJ enregistre toutes les commandes que l’on réalise avec son interface sur une image ; Ainsi, pas besoin de refaire la même procédure, il suffit alors d’appliquer la macro enregistrée sur la nouvelle image !
· Cliquer gauche sans relacher sur le nom de fichier correspondant à la photographie du gel à étudier « Plaque Elisa 1 », et le faire glisser jusqu'à la barre grise sous les pictogrammes de la barre d'outils :
Problématique
Réaliser une mesure d’absorbance sans spectrophotomètre : - sur des plaques de 96 puits pour des tests immunologique (type ELISA).
La procédure pour réaliser une lecture optique à l’aide d’ImageJ sans spectrophotomètre n’est pas compliquée, mais lorsqu’elle doit être réalisée sur une plaque de 96 puits, cela devient nettement plus contraignant surtout si cette analyse est à réaliser plusieurs fois par jour en laboratoire.
Ainsi, ImageJ propose à l’utilisateur de créer des Macros : Au moment où on le décide, ImageJ enregistre toutes les commandes que l’on réalise avec son interface sur une image ; Ainsi, pas besoin de refaire la même procédure, il suffit alors d’appliquer la macro enregistrée sur la nouvelle image !
· Cliquer gauche sans relacher sur le nom de fichier correspondant à la photographie du gel à étudier « Plaque Elisa 1 », et le faire glisser jusqu'à la barre grise sous les pictogrammes de la barre d'outils :
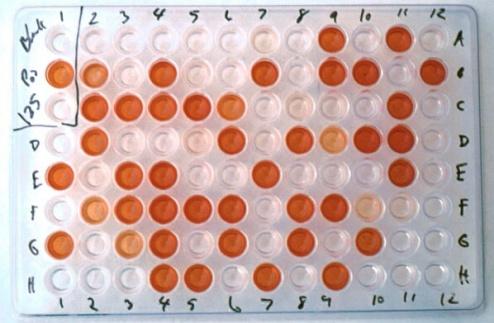
· Cliquez sur « Analyse »/ « Set mesurments » : décochez tout, sauf « Mean gray value » (valeur de gris moyenne). Puis OK ;
· Cliquez sur Plugins/Shortcuts/Create shortcut : une fenêtre « Create Shortcut » s’ouvre.
Au niveau de Command, choisir dans le menu déroulant : « Label », et en shortcut, choisir « F1 », puis cliquez sur OK.
· Dans la barre d’outils, cliquer sur Plugins/Macros/Record : Une fenêtre s’ouvre (dès lors, tout ce que l’on fait est enregistré !!) ;
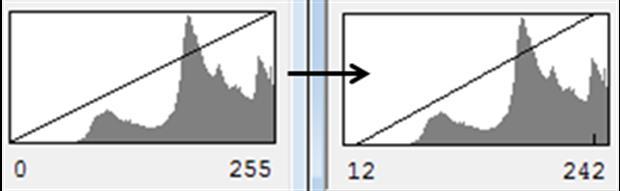
· Cliquer sur Image/Type/8-bit ; (Repérez en même temps comment s’écrit la Macro) ;
· Cliquer sur Image/Lookup Tables/Invert LUT : Le noir et blanc s’inverse ;
· Cliquer sur Image/Adjust/Brightness Contrast : et modifier uniquement le curseur Contrast de manière à obtenir les paramètres 12 et 242.
· Cliquer sur le deuxième icône « Oval » ;
· Placer le curseur de la souris à 10h de la périphérie du premier puits en haut à gauche, cliquez gauche en maintenant le clic et glissez en diagonale à 4h de la périphérie de ce premier puits : Relâcher le clic.
· Faites Ctrl C.
· * : Cliquez 1 fois au centre de l’image.
· Faites Crtl V : 1 cercle apparait au milieu de la photo. Placez le curseur de la souris au milieu de ce cercle, cliquez gauche en maintenant et faîtes glisser ce cercle sur le même premier puits en haut à gauche. Relâchez le clic.
· Faites Ctrl Z (le contenu du cercle s’efface ne laissant que son périmètre) ;
· Faites Ctrl M : un tableau de résultat s’ouvre avec le numéro du cercle et son niveau moyen (Mean) de gris ;
· Puis cliquez sur votre touche de shortcut (raccourcis : touche F1 en haut de votre clavier) de « Label » : le numéro de l’analyse s’inscrit sur l’image au niveau du puits étudié.
· Recommencez les étapes depuis l’ *, jusqu’à avoir marqué 6 cercles comme ci-dessous :
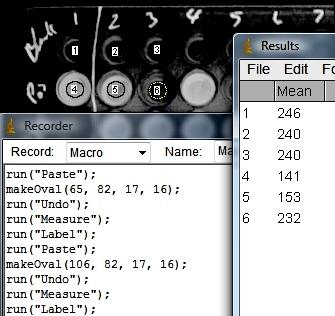
· Enfin, cliquer sur « Create » dans la fenêtre Recorder ; une fenêtre s’ouvre nommée « Macro.ijm » : c’est votre Macro finalisée.
· Dans cette même nouvelle fenêtre, faire File/Save as, et nommez là « MacroElisa6puits ».
· Fermez tout.
· Ouvrir cette fois ci, avec ImageJ, l’image nommée : « Plaque Elisa 2 ».
· Ouvrir le dossier où vous avez enregistré votre Macro, et faites glisser votre Macro vers la zone de lecture d’ImageJ : La fenêtre de vote macro s’ouvre.
· Dans cette fenêtre, faire Macros/Run Macro :
o L’image est alors travaillée et les puits analysés sont identifiés par des chiffres ;
o Les résultats des mesures de gris sont inscrits dans un tableau de résultats.
o Bien évidemment, ces résultats de ce tableau peut être récupéré dans Excel pour définir avec la fonction SI, si le puits est considéré comme positif ou négatif.
Remarque :
Vous avez pu remarquer dans la photo originale d’ELISA :
· qu’il n’y a pas que 2 couleurs extrêmes, mais on observe aussi des couleurs intermédiaires :
o Donc, il va falloir choisir une limite (ou un intervalle) de valeur de gris où l’on pourra conclure si le test est positif ou négatif.
· on peut aussi remarquer que la photographie a généré une sorte d’illumination à gauche de la plaque : ainsi deux puits ayant le même résultat mais se trouvant à un bord opposé de la plaque ne présenteront pas la même nuance de gris. Ceci explique en partie pourquoi les spectrophotomètres réalisent leurs mesures dans l’obscurité. Donc, si l’on veut réaliser cette analyse, en série, avec ImageJ, il faudra s’assurer que l’apport de lumière sur l’objet étudié est homogène sur toute sa surface (pas facile).
2) Dosage colorimétrique sur cuves spectrophotométriques : gamme d’étalonnage et essai
Ouvrez (faire glisser sur la barre d’ouverture d’ImageJ) la photo nommée « Gamme bleue et essai ».
Ouvrez (faire glisser sur la barre d’ouverture d’ImageJ) la photo nommée « Gamme bleue et essai ».


Cette photo représente, en cuve spectro, une gamme d’’étalonnage colorimétrique ainsi qu’un essai.
Sans spécifiquement produire une Macro, réalisez le même protocole ImageJ que précédemment en utilisant comme surface de sélection, non pas des cercles de diamètres égaux à ceux des puits des plaques, mais plutôt des rectangles couvrant au maximum le liquide contenu dans les cuves de spectro.
Ps : N’oubliez pas de passer en niveau de gris et d’inverser les noirs et blancs (si vous oubliez d’inverser, vous obtiendrez alors des niveaux de gris diminuant alors que la cuve est de plus en plus colorée… !).
Ps : faîtes en sorte que les résultats obtenus n’aient aucune décimale car le but est de réutiliser ces données dans un fichier Excel.

