
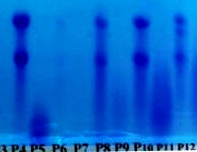

1. A la suite de manipulations indépendantes de biologie moléculaire (protocole ici) sur différentes espèces bactériennes, j'ai obtenu ce gel d'électrophorèse :
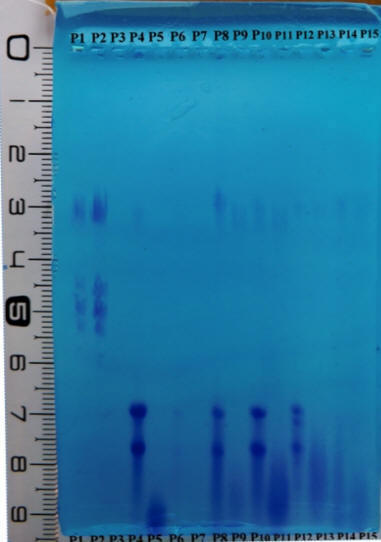
2. L'analyse densitométrique (logiciel ImageJ) de quelques pistes de cette électrophorèse donne les résultats suivant :
Sans faire d’analyse précise dans un premier temps, on peut faire quelques remarques sur cette partie basse du gel :
1) L’alternance des pistes paires et impaires (de la piste 4 à 15) présente toujours des résultats concordants dans cette partie basse du gel :
3) Les pistes 4, 8 et 10 semblent assez similaires ; Par contre, la piste 12 est clairement distincte !
C’est justement cette piste 12 qui m’a donné l’envie d’analyser plus en détail ce gel.
1) L’alternance des pistes paires et impaires (de la piste 4 à 15) présente toujours des résultats concordants dans cette partie basse du gel :
- pistes paires : deux grosses bandes bien visibles,
- pistes impaires : un smear plutôt bas.
3) Les pistes 4, 8 et 10 semblent assez similaires ; Par contre, la piste 12 est clairement distincte !
C’est justement cette piste 12 qui m’a donné l’envie d’analyser plus en détail ce gel.
Grâce au logiciel ImageJ, on peut placer les tailles des fragments (en paires de bases (pb)) sur le densitogramme pour tenter des comparaisons entre espèces bactériennes :
Ce qui est frappant avec les pics observés sur le densitogramme dans la partie basse du gel, dans les pistes paires, c’est que ces pics sont très élevés. D’ailleurs, il n’y a pas besoin du densitogramme pour déceler la présence abondante de ces fragments sur le gel ; on pourrait presque croire qu’il s’agit de produits de PCR tellement les tâches sont intenses.
Or il n’y a pas eu de PCR de réalisée. C’est donc que ces fragments nucléiques existent naturellement en un très grand nombre d’exemplaires dans la bactérie.
De si petites tailles, on pourrait penser à des petits plasmides ; Mais cette hypothèse ne colle pas avec l’observation du « double pic », sorte de couple majoritaire d’acides nucléiques, qui plus est, se retrouvant dans différentes espèces bactériennes.
Une hypothèse basée sur du génome phagique serait tout autant écartée avec les mêmes arguments que précédemment.
Ce double pic correspond donc à quelque chose de parfaitement bactérien hautement répété.
Un des complexes macromoléculaires les plus répétés des cellules sont les ribosomes. Ces derniers sont constitués d’ARNr.
Justement, les ARNr bactériens sont au nombre de 3, dont deux sont assez grands (l’ARNr 23S faisant 2904 nucléotides ; et l’ARNr 16S faisant 1541 nucléotides) et un dernier tout petit (l’ARNr 5S faisant 120 nucléotides).
Comme la stœchiométrie du ribosome est imposée : autant d’ARNr 23S que d’ARNr 16S, cela expliquerait l’observation de ce double pic.
Pour autant les tailles ne semblent pas coller…
Ce qui est frappant avec les pics observés sur le densitogramme dans la partie basse du gel, dans les pistes paires, c’est que ces pics sont très élevés. D’ailleurs, il n’y a pas besoin du densitogramme pour déceler la présence abondante de ces fragments sur le gel ; on pourrait presque croire qu’il s’agit de produits de PCR tellement les tâches sont intenses.
Or il n’y a pas eu de PCR de réalisée. C’est donc que ces fragments nucléiques existent naturellement en un très grand nombre d’exemplaires dans la bactérie.
De si petites tailles, on pourrait penser à des petits plasmides ; Mais cette hypothèse ne colle pas avec l’observation du « double pic », sorte de couple majoritaire d’acides nucléiques, qui plus est, se retrouvant dans différentes espèces bactériennes.
Une hypothèse basée sur du génome phagique serait tout autant écartée avec les mêmes arguments que précédemment.
Ce double pic correspond donc à quelque chose de parfaitement bactérien hautement répété.
Un des complexes macromoléculaires les plus répétés des cellules sont les ribosomes. Ces derniers sont constitués d’ARNr.
Justement, les ARNr bactériens sont au nombre de 3, dont deux sont assez grands (l’ARNr 23S faisant 2904 nucléotides ; et l’ARNr 16S faisant 1541 nucléotides) et un dernier tout petit (l’ARNr 5S faisant 120 nucléotides).
Comme la stœchiométrie du ribosome est imposée : autant d’ARNr 23S que d’ARNr 16S, cela expliquerait l’observation de ce double pic.
Pour autant les tailles ne semblent pas coller…
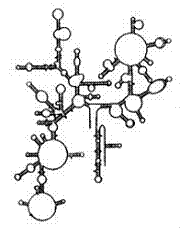
En fait, les tailles correspondent si on n’oublie pas de faire une petite conversion mentale : la caractéristique structurale de base des ARN est qu’ils sont monobrins. Ainsi leur taille est exprimée en nucléotides. Mais en réalité ils adoptent une structure secondaire complexe thermodynamiquement plus stable avec beaucoup de régions en double brin.
Dès lors, on peut imaginer que ces ARN se déplacent dans les mailles du gel non pas sous forme de monobrin mais plutôt sous une forme hybride majoritairement double brin (à gauche : l’ARNr 16S).
Dès lors, on peut imaginer que ces ARN se déplacent dans les mailles du gel non pas sous forme de monobrin mais plutôt sous une forme hybride majoritairement double brin (à gauche : l’ARNr 16S).
Ainsi, le double pic observé sur le gel correspond aux ARNr 23S et 16S ;
Normalement leur taille sont respectivement de 2904 et 1541 nucléotides chez E. coli, mais, dans le cas où ils se replieraient parfaitement en double brin, leur taille seraient alors de 1452pb et 770pb.
Le fait que l’on ne mesure pas exactement des tailles de 1452pb et 770pb pour ce double pic peut s’expliquer par l’absence de repliement total du monobrin en une structure double brin.
Voici un tableau récapitulatif des résultats obtenus (en pb) pour les 3 espèces :
Le fait que l’on ne mesure pas exactement des tailles de 1452pb et 770pb pour ce double pic peut s’expliquer par l’absence de repliement total du monobrin en une structure double brin.
Voici un tableau récapitulatif des résultats obtenus (en pb) pour les 3 espèces :
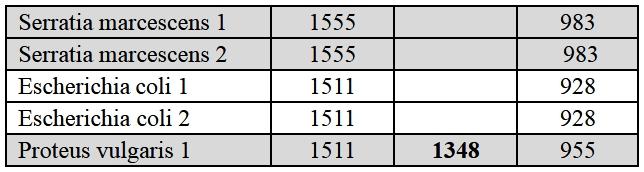
Ce tableau confirme la reproductibilité de cette expérience (5 extractions indépendantes)
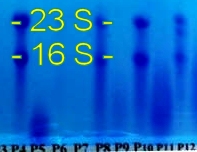
* Identification de la troisième bande chez Proteus vulgaris :
Lorsque dans une zone précise du gel, une espèce présente trois bandes électrophorétiques à contrario d’une autre espèce qui n’en présente que deux, il s’agit bien d’une distinction, soit de contenu ou d’expression, génétique.
C’est bien ce que l’on observe en piste 12. Il y a une troisième bande, clairement visible, juste en dessous de la bande de l’ARNr 23S.
Se pourrait-il que l’espèce Proteus vulgaris contienne trois grand ARNr dans ses ribosomes ?
En fait, il existe un processus cellulaire permettant d’obtenir un variant en taille d’un ARN : il s’agit de la maturation des ARN.
Rappelons tout d’abord que les gènes des ARNr 23S et 16S appartiennent à des opérons d’ARNr (E. coli possèdent 7 de ces opérons !) qui comprennent aussi le gène codant l’ARNr 5S (120 nucléotides) ainsi qu’un gène codant un ARNt (environ 80 nucléotides). L’ensemble de ces gènes sont transcrits en un transcrit primaire qui subira une maturation par une RNAse III libérant ainsi les différents ARN classiques.
Ce processus ne solutionne pas notre problème à moins que de nouveaux sites de liaison pour l’ARNase III soient apparus chez certaines espèces bactériennes.
Voici, d’après la littérature, à quoi ressemble le transcrit primaire correspondant à l’opéron d’ARNr chez E. coli (Journal of Bacteriology, 2000 February : 182 (4) : 1109 – 1117) :
Lorsque dans une zone précise du gel, une espèce présente trois bandes électrophorétiques à contrario d’une autre espèce qui n’en présente que deux, il s’agit bien d’une distinction, soit de contenu ou d’expression, génétique.
C’est bien ce que l’on observe en piste 12. Il y a une troisième bande, clairement visible, juste en dessous de la bande de l’ARNr 23S.
Se pourrait-il que l’espèce Proteus vulgaris contienne trois grand ARNr dans ses ribosomes ?
En fait, il existe un processus cellulaire permettant d’obtenir un variant en taille d’un ARN : il s’agit de la maturation des ARN.
Rappelons tout d’abord que les gènes des ARNr 23S et 16S appartiennent à des opérons d’ARNr (E. coli possèdent 7 de ces opérons !) qui comprennent aussi le gène codant l’ARNr 5S (120 nucléotides) ainsi qu’un gène codant un ARNt (environ 80 nucléotides). L’ensemble de ces gènes sont transcrits en un transcrit primaire qui subira une maturation par une RNAse III libérant ainsi les différents ARN classiques.
Ce processus ne solutionne pas notre problème à moins que de nouveaux sites de liaison pour l’ARNase III soient apparus chez certaines espèces bactériennes.
Voici, d’après la littérature, à quoi ressemble le transcrit primaire correspondant à l’opéron d’ARNr chez E. coli (Journal of Bacteriology, 2000 February : 182 (4) : 1109 – 1117) :
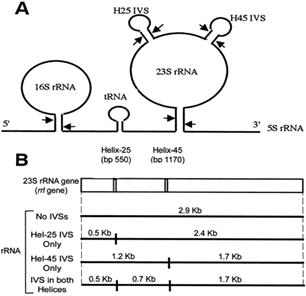
Fig. A : Structure secondaire du transcrit primaire contenant l’ARNr 16S, un ARNt, l’ARNr 23S et l’ARNr 5S ; Les flèches indiquent des sites d’hydrolyse par l’ARNase III.
L’ARNr 23S se présente sous la forme d’une grande structure en épingle surmontée de deux petites structures en épingle nommées H25 IVS et H45 IVS. Selon les espèces bactériennes, les sites de clivages pour l’ARNase III à la base des petites épingles sont oui ou non présents.
Fig. B : Tailles des différents ARNr 23S possibles : 4 possibilités : 1) aucun site de coupure sous les IVS, 2) et 3) 1 seul site de coupure sous H25 ou H45, et 4) 2 sites de coupure sous H25 et H45.
L’ARNr 23S se présente sous la forme d’une grande structure en épingle surmontée de deux petites structures en épingle nommées H25 IVS et H45 IVS. Selon les espèces bactériennes, les sites de clivages pour l’ARNase III à la base des petites épingles sont oui ou non présents.
Fig. B : Tailles des différents ARNr 23S possibles : 4 possibilités : 1) aucun site de coupure sous les IVS, 2) et 3) 1 seul site de coupure sous H25 ou H45, et 4) 2 sites de coupure sous H25 et H45.
D’après notre expérience, on en déduit que Proteus vulgaris subi un clivage supplémentaire au niveau de la structure en épingle H25 IVS générant ainsi de l’ARNr 23S de grande taille ainsi que de l’ARNr 23S de taille légèrement inférieur.
Conclusion :
Je n’ai pas réussi à extraire des génomes bactériens complets. Des lors, la simulation sous Excel de la digestion enzymatique de ces génomes est pour le moment non utilisable (à tester à l’aide de kits d’extraction génomique).
Par chance, notre laboratoire ne possédait pas de RNAse. Ainsi on a pu observer que certaines espèces bactériennes présentaient naturellement des distinctions génétiques de leur ARNr 23S. Selon l’article anglais cité ci-dessus, les Proteus, les Providencia mais aussi les Salmonella présenteraient des variants de taille de leurs ARNr 23S.
Il est donc possible, par une simple extraction nucléique suivi d’une électrophorèse, de distinguer génétiquement certaines espèces bactériennes. Le signal étudié, l’ARNr 23S sera fort puisqu’il est produit pour l’élaboration du complexe macromoléculaire le plus représenté de toutes les cellules : le ribosome.
Conclusion :
Je n’ai pas réussi à extraire des génomes bactériens complets. Des lors, la simulation sous Excel de la digestion enzymatique de ces génomes est pour le moment non utilisable (à tester à l’aide de kits d’extraction génomique).
Par chance, notre laboratoire ne possédait pas de RNAse. Ainsi on a pu observer que certaines espèces bactériennes présentaient naturellement des distinctions génétiques de leur ARNr 23S. Selon l’article anglais cité ci-dessus, les Proteus, les Providencia mais aussi les Salmonella présenteraient des variants de taille de leurs ARNr 23S.
Il est donc possible, par une simple extraction nucléique suivi d’une électrophorèse, de distinguer génétiquement certaines espèces bactériennes. Le signal étudié, l’ARNr 23S sera fort puisqu’il est produit pour l’élaboration du complexe macromoléculaire le plus représenté de toutes les cellules : le ribosome.

